|
| |
| Residual viremia quantification challenges |
|
| Francesco R. Simonetti, Divisione di Infettivologia, Azienda Ospedaliera Luigi Sacco, MIlano |
| |
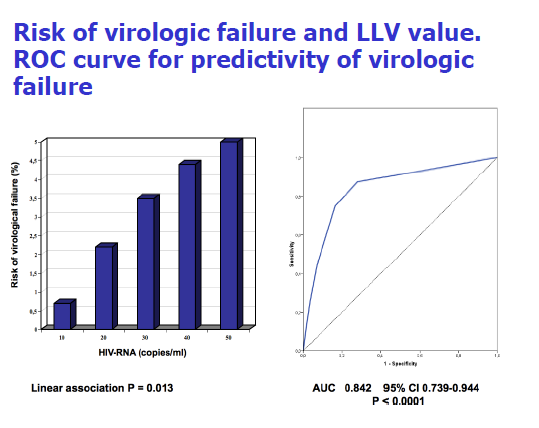 |
| Cologni G, #348 |
|
| |
| Patogenesi -
La viremia residua correla con i livelli di HIV-DNA totale (Cossarini F, #350) e con il numero di unità virali infettanti (Gandhi R, #352) mentre non si modifica nei pazienti stabilmente sotto HAART sottoposti a regimi chemio-ablativi o ad allotrapianto (Cillo A, #353, Cillo A, #154). |
| |
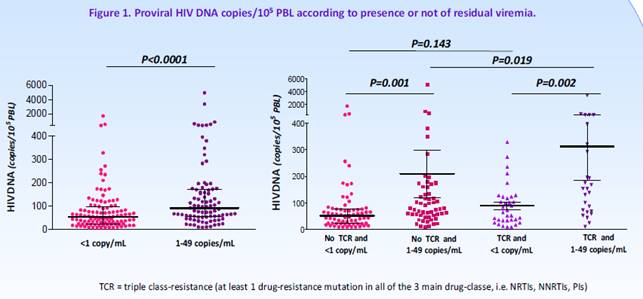 |
| Cossarini F, #350 |
|
| |
È stato possibile, mediante single copy assay (SCA), caratterizzare l’andamento trifasico della cinetica di calo dopo l’introduzione della terapia (rispettivamente 1.5 giorni, 10 giorni e 62 settimane; Andrade A, #672).
Ancora dibattuto, invece, se nei pazienti soppressi vi sia evoluzione delle quasi-specie con rischio di selezione di resistenza o di switch nel tropismo (Brennan T, #345, Raymond S, #346, Delaguerre C, #347, Josefsson L, #361). |
|
| Metodologia - È stata presentata una nuova droplet digital PCR che è in grado di amplificare singole copie di HIV-RNA all’interno di 20,000 micelle lipidiche in sospensione (Anderson E, #679). È stata descritta una metodica innovativa, seppur poco sensibile, che permette di quantificare RNA di qualunque clade di HIV-1 (M e O) così come di SIVcpz/gor (Etienne L, #680). L’assenza di segnale (HIV-RNA non determinato) dei saggi usati in pratica clinica può essere usata come alternativa efficace ai più costosi SCA (Hoen B, #75). |