| Resistenze |
|
|
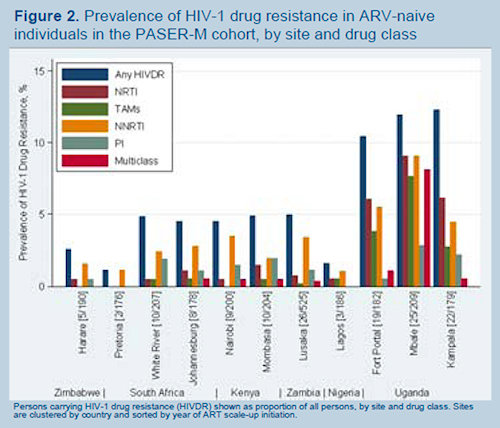
| Tale dato merita attenzione anche se la metodologia adottata richiede studi di conferma epidemiologica. Inoltre, studi effettuati con metodologie diverse, confermano che la maggior parte dei paesi Africani presenta una prevalenza di TDR<5% (#619, #620, #622), mentre paesi selezionati come Uganda, Vietnam, Messico e Brasile presentano prevalenze tra il 5-15%, con prevalenza di TDR a NRTI in America latina ed NNRTI in Africa (#621, #625). Inoltre, la prevalenza di TDR correla, come atteso, con il tempo trascorso dall’implementazione dei programmi ART (#622). |
| |
 |
| |
| I programmi di sorveglianza OMS delle ADR in coorti di pazienti ad 1 anno dall’inizio della ART (15 studi in 5 paesi; #52) ha mostrato un 10% di fallimento virologico (VL>1000, analisi on treatment): nei falliti le sequenze mostravano 65% di ADR agli NNRTI e 52% ai NRTI con poche TAM e compromissione del TDF (essenziale per la seconda linea con PI/r in questi paesi) in meno del 10% dei test. Il dato va interpretato con cautela, alla luce del fatto che si tratta naturalmente di fallimenti “precoci”, specialmente per paesi dove, in mancanza di monitoraggio virologico, il passaggio alla seconda linea è molto più tardivo. Altri studi hanno infatti dimostrato come le ADR al fallimento con monitoraggio dei soli CD4 sono molto più estese, anche se le resistenze significative al TDF (dopo fallimento con analoghi timidinici) non superavano il 32% (#616), mentre le TAM tendevano ad aumentare nel secondo anno di ART, con presenza di >=3TAM in >50% a 2 anni (#618). |
|
| La mutazione prevalente di TDR ed ADR nei paesi in via di sviluppo appare essere la K103N che, in considerazione dell’ampiezza delle popolazioni, si candida ad essere la mutazione principale di resistenza a livello globale. |
|
| In un’analisi dei primi fallimenti virologici in Francia, il confronto tra TDF/FTC e TDF+3TC con EFV o con PI/r, mostrava una percentuale superiore di M184V/I con 3TC rispetto a FTC (51% vs 24% con EFV e 22% vs 11% con PI/r) ma nessuna differenza nelle prevalenze di resistenza ai NNRTI o PI (#617). |
|
| Ulteriori evidenze sono emerse circa le implicazioni cliniche dei nuovi test del tropismo. Un’analisi estesa dei campioni dei trials Motivate e 1029 ha dimostrato un’ottima capacità discriminativa della risposta virologica (di ceppi R5 vs non-R5) a 8 settimane con i test genotipico del tropismo con sequenziamento standard della gp120 V3 (interpretato con geno2pheno), ESTA e sequenziamento “ultradeep”. Gli ultimi due mostravano un lievissimo vantaggio in termini di capacità discriminatoria della risposta (#666). |
|
| Altri hanno dimostrato come il livello di false positive rate riportato dal geno2pheno concordasse con la percentuale di quasi specie X4 nel pool virale plasmatico (#667). |
|
| Molto rilevante dal punto di vista clinico il rilievo di 2 studi circa la rilevazione genotipica del tropismo su DNA virale in PBMC che conferma una associazione con la risposta virologica (#668) e con il tropismo rilevato su RNA virale plasmatico prima della soppressione virologica (#669). Questi dati incoraggiano la possibilità di testare il tropismo sul DNA virale in soggetti virosoppressi allo scopo di poter impiegare gli antagonisti di CCR5 anche in tale contesto. |
|
| Per quanto attiene la classe dei NNRTI, una sottoanalisi dello studio SMART ha dimostrato come dopo interruzione degli NNRTI, la risposta virologica alla ripresa di NNRTI fosse ridotta in un sottogruppo di pazienti con maggiore esposizione alla ART, lenta eliminazione degli NNRTI e con mutazioni a 2 classi (#596). |
|
| La interpretazione della resistenza ad etravirina rimane discordante tra sistemi esperti: solo le interpretazioni di resistenza secondo ANRS e lo score Tibotec sono significativamente associate alla risposta ad ETR in Eurosida (#598), mentre la presenza di K103N sembrerebbe associata ad una maggiore risposta virologica. Per i PI due studi hanno dimostrato come diversi polimorfismi della regione gag possano ridurre la barriera genetica del darunavir in vitro (I436S/T) (#603) e la risposta virologica al farmaco in vivo (A431V) (#604). |
|
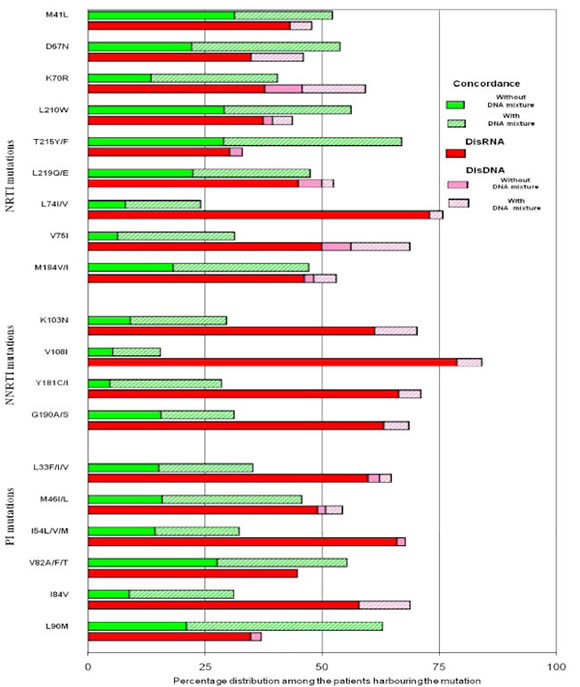
| Infine, uno studio (#613) ha analizzato la presenza di resistenze in HIV DNA in 151 pazienti nei quali non era stato possibile sequenziare l’HIV RNA plasmatico per viremia bassa o soppressa e che avevano test di resistenza storici su HIV RNA (>3 in media). |
| |
 |
| |
La concordanza tra mutazioni di resistenza nel DNA e quelle precedentemente rilevate nell’RNA era di appena il 46,7% per NRTI, 26.3% per NNRTI e 43.7% per PI. La maggior parte delle discordanze era dovuta alla mancata capacità di rilevare le resistenze nel DNA, mentre l’opposto (resistenze nel DNA ma non nei precedenti RNA) appariva raro (<9%). In pazienti non sequenziabili il genotipo storico appare quindi più utile per le decisioni terapeutiche rispetto al genotipo su DNA virale, anche se questo risultato deve ancora essere corroborato dai dati di risposta terapeutica. |